![]()
![]() - ⇑
- ⇑

¡Necesitas Javascript para poder usar este menú adecuadamente!
He estudiado evolución en el ADN y proteínas, y malaria.

¡Necesitas Javascript para poder usar este menú adecuadamente!

Mi meta es desarrollar métodos estadísticos, modelos probabilisticos, y algoritmos para problemas en la biología computacional. Estoy particularmente interesado en enfermedades y evolución humana, incluyendo el estudio de genética de poblaciones y secuencias de proteína. Analizo conjuntos de datos grandes y afronto problemas coun una variedad de herramientas de la estadística y ciencias computacionales, balanceando la precisión con el rendimiento. Ejemplos incluyen tomar en cuenta el parentesco genómico para mejor asociar genotipos a enfermedades, mejorar las predicciones de dominios en bases datos grandes de proteínas, modelar puntajes psiquiátricos en ensayos clínicos, y expander anotaciones funcionales en parásitos de la malaria.
Soy un Profesor Asistente en el Departamento de Bioestadística y Bioinformática en la Universidad de Duke. Mi grupo de investigación, el Laboratorio Ochoa, es parte del Centro de Genética y Genómica Estadística de Duke.
Fuí un investigador de postdoctorado en el laboratorio de John D Storey en la Universidad de Princeton hasta 2018. Recibí my doctorado en Biología Molecular en el 2013 de la Universidad de Princeton, coasesorado por Mona Singh y Manuel Llinás. En el 2006 terminé la carrera doble en biología y matemáticas en el Instituto de Tecnología de Massachusetts. Vea mi CV (PDF).

2021-01-19. Alejandro Ochoa, John D Storey. Estimating FST and kinship for arbitrary population structures. PLoS Genet. 17(1) e1009241. PubMed. PubMed Central. Artículo. bioRxiv 2016-10-27.
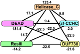
Alejandro Ochoa, John D Storey. FST and kinship for arbitrary population structures I: Generalized definitions. bioRxiv 2016-10-27.

2017-04-12. Alejandro Ochoa, Mona Singh. Domain prediction with probabilistic directional context. Bioinf. 33(16) 2471-8. PubMed. PubMed Central. Artículo. bioRxiv 2016-12-14.
Programa: dPUC2

2015-11-17. Alejandro Ochoa, John D Storey, Manuel Llinás, Mona Singh. Beyond the E-value: stratified statistics for protein domain prediction. PLoS Comput Biol. 11 e1004509. PubMed. PubMed Central. Artículo. arXiv 2014-09-23.
Programas: DomStratStats y RandProt
2011-03-31. Alejandro Ochoa, Manuel Llinás, Mona Singh. Using context to improve protein domain identification. BMC Bioinformatics. 12(1) 90. PubMed. PubMed Central. Artículo.
Programa: dPUC1

2016-01-27. Simon A Cobbold, Joana M Santos, Alejandro Ochoa, David H Perlman, Manuel Llinás. Proteome-wide analysis reveals widespread lysine acetylation of major protein complexes in the malaria parasite. Sci Rep. 2016;6:19722. PubMed. PubMed Central. Artículo.
.

2013-11-13. Moriah L Szpara, Derek Gatherer, Alejandro Ochoa, Benjamin Greenbaum, Aidan Dolan, Rory J Bowden, Lynn W Enquist, Matthieu Legendre, Andrew J Davison. Evolution and diversity in human herpes simplex virus genomes. J Virol. 88:1209-27. PubMed. PubMed Central. Artículo.

2007-11-01. Gevorg Grigoryan, Alejandro Ochoa, Amy E Keating. Computing van der Waals energies in the context of the rotamer approximation. Proteins. 68(4) 863-78. PubMed. Artículo.
.